摘 要 / Abstract
蛋白質(zhì)組學(xué)研究是繼基因組學(xué)研究興起之后的又一新興領(lǐng)域���,為相關(guān)科學(xué)研究提供了全新的思路和技術(shù)���,已在藥品和生物制品領(lǐng)域得到了廣泛應(yīng)用��。近年來(lái)��,蛋白質(zhì)組學(xué)技術(shù)開(kāi)始逐漸被應(yīng)用于食品和化妝品檢測(cè)領(lǐng)域��,有望為開(kāi)展食品和化妝品安全���、質(zhì)量控制等研究提供有力手段。本文主要綜述了已應(yīng)用蛋白質(zhì)組學(xué)技術(shù)進(jìn)行研究的食品和化妝品種類(lèi)���,并介紹其技術(shù)流程特點(diǎn)���,對(duì)分析流程中亟待解決的技術(shù)難點(diǎn)進(jìn)行了剖析。蛋白質(zhì)組學(xué)技術(shù)具有其他傳統(tǒng)方法不可取代的高準(zhǔn)確性��、高通量的檢測(cè)優(yōu)勢(shì)�����,同時(shí)需要注意���,其在食品和化妝品檢測(cè)領(lǐng)域的方法回收率低���、標(biāo)準(zhǔn)物質(zhì)研制滯后等主要問(wèn)題仍有待克服。
Proteomics research, emerging after the rise of genomics, provides new ideas and techniques for related scientific research and has found ample applications in drugs and biological products. In recent years, proteomic techniques have gradually been applied to the field of food and cosmetics detection, offering a powerful means for research on the safety and quality control of these products. This paper reviews the types of foods and cosmetics studied using proteomics techniques, introduces the characteristics of their technical processes, and analyzes the technical difficulties in the analysis process. Proteomics technique boasts high accuracy and throughput, which are irreplaceable compared to traditional methods. However, challenges such as low method recovery and the lagging development of reference materials still need to be overcome in the field of food and cosmetics detection.
關(guān) 鍵 詞 / Key words
蛋白質(zhì)組學(xué)��;食品�����;化妝品; 高準(zhǔn)確性�����;高通量
proteomics; food; cosmetics; high accuracy; high throughput
食品安全是人類(lèi)面臨的一個(gè)重大挑戰(zhàn)���,人們對(duì)食品的關(guān)注已經(jīng)不僅僅局限于其營(yíng)養(yǎng)價(jià)值��,還越來(lái)越重視其安全性���。如何全面評(píng)價(jià)食品質(zhì)量,實(shí)現(xiàn)對(duì)食品質(zhì)量全過(guò)程的把控頗為重要[1]���。我國(guó)《食品安全法》[2] 的實(shí)施為國(guó)內(nèi)食品安全提供了有力保障��。食品質(zhì)量受原材料��、生產(chǎn)加工���、儲(chǔ)存運(yùn)輸?shù)榷喾N因素的影響��,相關(guān)質(zhì)量問(wèn)題包括含有傳統(tǒng)的化學(xué)危害物��、致病菌���,以及以次充好、以劣質(zhì)原材料生產(chǎn)高價(jià)值產(chǎn)品��、產(chǎn)地造假等[3-6]��。
隨著人們生活質(zhì)量的提高�����,化妝品日漸成為生活必需品��。根據(jù)中國(guó)香料香精化妝品工業(yè)協(xié)會(huì)不完全統(tǒng)計(jì)�����,2020 年我國(guó)化妝品生產(chǎn)企業(yè)主營(yíng)業(yè)務(wù)收入達(dá)3950億元[7]。隨著化妝品生產(chǎn)工藝的發(fā)展和銷(xiāo)量的高速增長(zhǎng)���,產(chǎn)品質(zhì)量和安全受到越來(lái)越多的關(guān)注。同時(shí)��,隨著更多化妝品新材料被發(fā)現(xiàn)�����,如蛋白質(zhì)和多肽類(lèi)物質(zhì)�����,在提高產(chǎn)品質(zhì)量和功效的同時(shí)���,相關(guān)安全問(wèn)題也在增加[8]�����。為保障化妝品質(zhì)量��,加強(qiáng)化妝品質(zhì)量安全監(jiān)管��,我國(guó)發(fā)布了《化妝品安全技術(shù)規(guī)范(2015 年版)》[9]�����,明確了化妝品禁限用組分��。
蛋白質(zhì)分別作為主要營(yíng)養(yǎng)成分和新型化妝品材料存在于食品和相關(guān)化妝品中�����,參與決定了食品和化妝品質(zhì)量��。傳統(tǒng)的蛋白質(zhì)研究方法有酶聯(lián)免疫吸附測(cè)定法(ELISA 法)���、Folin- 酚試劑法(也稱(chēng)為L(zhǎng)owry 法)�����、考馬斯亮藍(lán)法(Braford 法)和凱氏定氮法等[10-12]��。上述方法只能用于總蛋白含量測(cè)定�����,無(wú)法對(duì)具體的營(yíng)養(yǎng)蛋白或者過(guò)敏原進(jìn)行定性定量檢測(cè)���,且研究通量低�����,具有局限性���。蛋白質(zhì)組學(xué)是繼基因組學(xué)研究興起之后的又一新興領(lǐng)域,與傳統(tǒng)蛋白質(zhì)研究方法相比��,能夠用于蛋白質(zhì)的鑒定���、定量、定位��、蛋白結(jié)構(gòu)修飾分析和蛋白質(zhì)- 蛋白質(zhì)相互作用研究[13-15]�����,且研究通量高�����, 研究范圍更加完整��。因此��,蛋白質(zhì)組學(xué)技術(shù)為相關(guān)科學(xué)研究提供了全新的思路和技術(shù),已在藥品和生物制品領(lǐng)域得到廣泛應(yīng)用[16-17]��。近年來(lái)��,蛋白質(zhì)組學(xué)逐漸被應(yīng)用于食品和化妝品等領(lǐng)域��,如品質(zhì)評(píng)價(jià)��、風(fēng)險(xiǎn)發(fā)現(xiàn)�����、產(chǎn)品真實(shí)性檢驗(yàn)�����、產(chǎn)品摻雜摻假檢測(cè)和產(chǎn)品功效評(píng)價(jià)等[18-19]��,其在食品和化妝品領(lǐng)域的應(yīng)用范圍和解決問(wèn)題如表1 所示�����。
1���、蛋白質(zhì)組學(xué)技術(shù)介紹
經(jīng)過(guò)數(shù)十年的發(fā)展���,基于質(zhì)譜的蛋白質(zhì)組學(xué)技術(shù)已形成了一套較為完整的分析流程���,常規(guī)分析步驟主要包括:樣品前處理、分離��、質(zhì)譜檢測(cè)和質(zhì)譜數(shù)據(jù)分析���。其中���,樣品前處理是整個(gè)蛋白質(zhì)組學(xué)分析流程中的關(guān)鍵步驟��,影響分析穩(wěn)定性�����、準(zhǔn)確性和靈敏度[20]�����。傳統(tǒng)的樣品制備方法步驟多���、耗時(shí)長(zhǎng)�����,且大部分依靠手工操作�����,不可避免地存在人為誤差���。值得關(guān)注的是��,自動(dòng)化樣品制備平臺(tái)的相繼出現(xiàn)�����,如貝爾曼庫(kù)爾特的Biomek® NXP 自動(dòng)化工作站[21]和賽默飛的全自動(dòng)磁珠提取純化系統(tǒng)KingFisher Flex 等���, 有助于提高生物標(biāo)志物候選物的通量和可重現(xiàn)的定量,在臨床��、藥品和生物制品領(lǐng)域已經(jīng)有所應(yīng)用[22-23]���。需要注意的是��,在食品樣品制備領(lǐng)域���,高通量樣品制備技術(shù)尚未得到應(yīng)用��,大多仍依靠手工操作���,其步驟煩瑣、樣品回收率偏低�����,有待進(jìn)一步優(yōu)化�����。
2�����、蛋白質(zhì)組學(xué)技術(shù)在食品質(zhì)量檢測(cè)領(lǐng)域的應(yīng)用
蛋白質(zhì)組學(xué)技術(shù)的發(fā)展為食品相關(guān)研究打開(kāi)了新思路���,不僅可以用于蛋白質(zhì)種類(lèi)鑒定,還可用于蛋白質(zhì)含量檢測(cè)��,以及分析不同物種、產(chǎn)地���、生長(zhǎng)階段的食品蛋白質(zhì)組分和食品加工過(guò)程蛋白質(zhì)組變化等�����。目前��,蛋白質(zhì)組學(xué)技術(shù)已在食品的諸多品類(lèi)中有應(yīng)用���,如農(nóng)產(chǎn)品、海產(chǎn)品���、乳制品�����、肉類(lèi)�����、微生物類(lèi)以及食品過(guò)敏原等(表2)��。
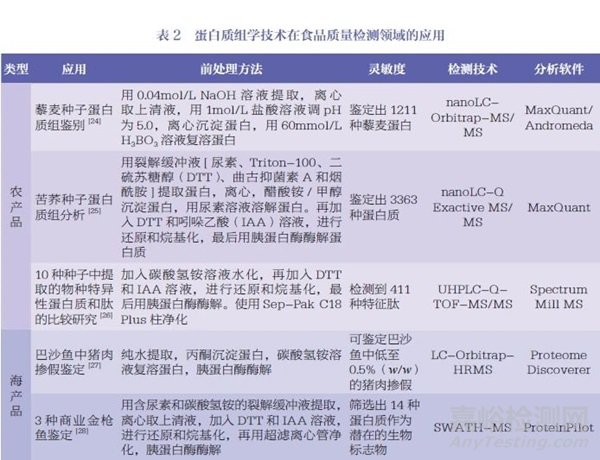
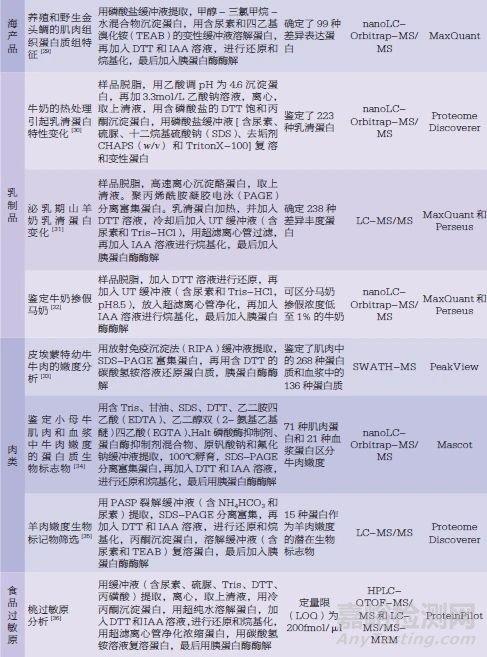

蛋白質(zhì)組學(xué)中基于質(zhì)譜的分析方法是食品質(zhì)量分析領(lǐng)域中發(fā)展較快的方法�����, 研究流程分為Bottom-up 型和Top-down型�����。前者又稱(chēng)為鳥(niǎo)槍法��,該技術(shù)流程一般分為以下幾步:樣品制備�����,即從樣品中分離凈化目標(biāo)物��;蛋白質(zhì)酶解���,將蛋白質(zhì)酶解為肽段�����;利用色譜技術(shù)對(duì)肽段進(jìn)行分離,隨后進(jìn)入質(zhì)譜檢測(cè)��。相較于Bottom-up 技術(shù)���,Top-down 技術(shù)的不同之處在于蛋白質(zhì)未經(jīng)酶解直接進(jìn)入質(zhì)譜���,直接被裂解為肽段進(jìn)行分析��。該方法成本高���,目前應(yīng)用較少。蛋白質(zhì)組學(xué)分析流程見(jiàn)圖1�����。
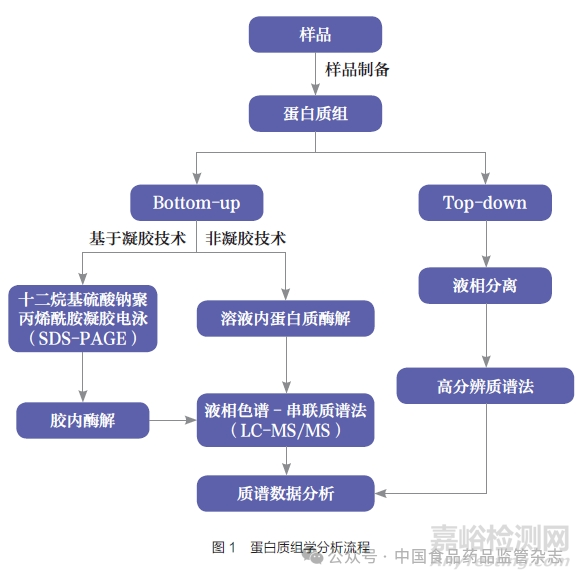
3�����、蛋白質(zhì)組學(xué)技術(shù)在化妝品質(zhì)量檢測(cè)領(lǐng)域的應(yīng)用
蛋白質(zhì)和多肽類(lèi)原料在化妝品生產(chǎn)中的應(yīng)用日益增多��,如膠原蛋白�����、蠶絲蛋白�����、牛奶提取蛋白、彈性蛋白�����、角蛋白�����、小麥水解蛋白等在各類(lèi)化妝品中已有應(yīng)用�����。我國(guó)《已使用化妝品原料目錄(2021 年版)》[42] 中屬于蛋白質(zhì)類(lèi)和肽類(lèi)的相關(guān)原料已有213種�����。蛋白質(zhì)類(lèi)原料的應(yīng)用可以使化妝品具有抗衰老��、美白�����、保濕等功效[43]���,故該類(lèi)原料已成為各大化妝品品牌的競(jìng)爭(zhēng)亮點(diǎn)和開(kāi)展基礎(chǔ)研究的重點(diǎn)關(guān)注對(duì)象��。但由于有些內(nèi)源性蛋白質(zhì)或多肽具有相對(duì)較高的生物活性�����,可能具有致敏性���,實(shí)際應(yīng)用過(guò)程中應(yīng)予以重點(diǎn)關(guān)注[44]。筆者查閱國(guó)內(nèi)外相關(guān)文獻(xiàn)發(fā)現(xiàn)��,當(dāng)前已有基于質(zhì)譜的蛋白質(zhì)組學(xué)技術(shù)應(yīng)用于化妝品中常見(jiàn)蛋白質(zhì)原料檢測(cè)方面的研究��,但對(duì)于實(shí)際化妝品樣品中蛋白質(zhì)應(yīng)用檢測(cè)的蛋白組學(xué)技術(shù)研究尚未有報(bào)道���。
3.1 化妝品中動(dòng)物來(lái)源蛋白質(zhì)原料的分析特點(diǎn)
目前�����, 應(yīng)用于化妝品的蛋白質(zhì)類(lèi)和肽類(lèi)原料已有213 種�����,其中常用的動(dòng)物來(lái)源蛋白質(zhì)主要有膠原蛋白��、角蛋白���、骨膠原�����、胎盤(pán)蛋白( 動(dòng)物)�����、彈性蛋白�����、牛奶蛋白等�����。該類(lèi)原料的使用對(duì)皮膚具有一定功能性作用��, 在化妝品中應(yīng)用廣泛�����。然而��, 一些不法商家夸大宣稱(chēng)或不恰當(dāng)宣傳其功效���,從而誤導(dǎo)消費(fèi)者��。此外,該類(lèi)原料價(jià)格相對(duì)較高�����,部分原料對(duì)儲(chǔ)存條件有一定要求��。因此, 應(yīng)當(dāng)對(duì)該類(lèi)原料使用的合規(guī)性多加關(guān)注�����, 如實(shí)際產(chǎn)品與配方的一致性��、原料的保管和來(lái)源等��。
膠原蛋白是最常用于化妝品的動(dòng)物來(lái)源蛋白質(zhì)原料��,因其具有保濕�����、再生和成膜特性�����,有助于皮膚保持適當(dāng)?shù)乃趾?�,從而滋?rùn)和軟化皮膚,還可防止機(jī)械損傷引起的皮膚和頭發(fā)損傷[45]�����。因此���,膠原蛋白被廣泛應(yīng)用于美容領(lǐng)域�����。目前,膠原蛋白主要從哺乳動(dòng)物和魚(yú)皮中提取[46]�����,但哺乳動(dòng)物來(lái)源的膠原蛋白具有疾病傳染風(fēng)險(xiǎn)���,魚(yú)皮中提取的膠原蛋白則具有獨(dú)特的應(yīng)用優(yōu)勢(shì)���。研究表明,海洋膠原蛋白中富含Ⅰ型膠原蛋白[47]���,人皮膚中主要的膠原蛋白類(lèi)型也是Ⅰ型[48]��,且沒(méi)有疾病傳播風(fēng)險(xiǎn)�����,是化妝品領(lǐng)域較為理想的膠原蛋白��。因此�����,有必要針對(duì)相關(guān)產(chǎn)品中的膠原蛋白類(lèi)型及來(lái)源進(jìn)行檢測(cè)��。例如���,有研究者利用離子阱質(zhì)譜儀鑒定Ⅰ型和Ⅲ型膠原蛋白的標(biāo)記肽���,使用三重四極桿質(zhì)譜儀進(jìn)行含量測(cè)定[49]。還有研究者使用快速簡(jiǎn)便的液相色譜- 串聯(lián)質(zhì)譜法(LC-MS/MS)鑒定膠原蛋白標(biāo)記肽�����,該方法可以同時(shí)檢測(cè)6 種動(dòng)物來(lái)源的膠原蛋白[50]���?�?傊?����,對(duì)于化妝品中的動(dòng)物來(lái)源蛋白質(zhì)原料��,注重分析其來(lái)源和類(lèi)型���。
3.2 化妝品中植物來(lái)源蛋白質(zhì)原料的分析特點(diǎn)
植物蛋白對(duì)人體的皮膚具有較好的親和力�����,可以提高表皮保濕性能,同時(shí)具有成膜性[51]��。植物蛋白含人體8 種必需氨基酸��,能促進(jìn)表皮生長(zhǎng)發(fā)育�����、修復(fù)組織�����、抗衰老、去角質(zhì)等���。植物來(lái)源的蛋白質(zhì)類(lèi)和肽類(lèi)原料主要用于皮膚或頭發(fā)調(diào)理劑等個(gè)人護(hù)理產(chǎn)品�����。應(yīng)用于化妝品的植物蛋白主要分為2 種:植物蛋白水解物和水解物的衍生物���。其中,水解植物蛋白在個(gè)人護(hù)理產(chǎn)品中的用途報(bào)道最多���, 共有140 余種配方���,約有一半用于免洗產(chǎn)品,且涉及種類(lèi)繁多��,如大豆蛋白��、小麥蛋白�����、燕麥蛋白��、杏仁提取蛋白、玉米蛋白及其水解蛋白等[52]�����。值得關(guān)注的是��,歐盟委員會(huì)已經(jīng)對(duì)歐盟化妝品法規(guī)(EC)No.1223/2009 中的“化妝品限用物質(zhì)清單”(附錄Ⅲ)進(jìn)行了修訂�����,限制了花生油和水解小麥蛋白在化妝品中的使用�����,規(guī)定化妝品中使用的花生油提取物或衍生物的蛋白最大濃度設(shè)定為0.5μg/ml��,水解小麥蛋白中多肽的最大分子量平均值限制為3.5kDa[53]���。總之��,對(duì)化妝品中植物來(lái)源的蛋白質(zhì)類(lèi)和肽類(lèi)原料的關(guān)注重點(diǎn)是對(duì)具有生物活性的該類(lèi)原料進(jìn)行鑒定及含量檢測(cè)�����。
有研究者利用質(zhì)譜法對(duì)玉米酒糟的蛋白水解物進(jìn)行分析鑒定,鑒定出APLA��、PLFP��、LFLP��、LPPYL�����、PLYPLP�����、NDWHTGPL�����、LPPYLPS��、GSPFLGQ�����、SWQQPIVGG 等多個(gè)抗氧化活性肽[54]�����。也有研究者通過(guò)使用質(zhì)譜法確定了3 個(gè)表型組(WDEIA、AD�����、PR)的特定致敏譜��,并通過(guò)LC-MS/MS 確定了幾種小麥過(guò)敏原[55]�����。還有研究者開(kāi)發(fā)了一種外標(biāo)肽段定量的高靈敏LCMS/MS 檢測(cè)方法���,用于定量分析大豆樣品中Gly m 5.0101 的含量��, 通過(guò)對(duì)Gly m 5.0101 的特異性標(biāo)記肽NPFLFGSNR 定量�����,檢測(cè)各種大豆種子和豆粕中的痕量Gly m 5.0101[56]���。
4�����、蛋白質(zhì)組學(xué)技術(shù)用于食品和化妝品檢測(cè)相關(guān)研究面臨的問(wèn)題與建議
4.1 研究開(kāi)發(fā)過(guò)程中的問(wèn)題與建議
基于質(zhì)譜的蛋白質(zhì)組學(xué)技術(shù)發(fā)展至今,從樣品制備���、蛋白質(zhì)組質(zhì)譜數(shù)據(jù)采集到質(zhì)譜數(shù)據(jù)分析都取得了快速發(fā)展���。其中,樣品制備是相關(guān)工作流程中的關(guān)鍵步驟�����,影響分析的穩(wěn)健性��、準(zhǔn)確性和靈敏度���,分析步驟主要包括細(xì)胞裂解���、蛋白質(zhì)提取、分離���、酶消化和脫鹽[57]��。蛋白質(zhì)組學(xué)技術(shù)在食品領(lǐng)域應(yīng)用面臨的一大問(wèn)題是蛋白質(zhì)回收率普遍偏低�����,如何有效提高提取效率是提高該類(lèi)方法回收率的關(guān)鍵�����。在提取食品中蛋白質(zhì)時(shí)�����,大多通過(guò)使用裂解緩沖液提高提取效率�����。裂解緩沖液含有尿素��、硫脲���、十二烷基硫酸鈉(SDS)���、二硫蘇糖醇(DTT)、乙二胺四乙酸(EDTA)��、乙二醇雙(2- 氨基乙基醚)四乙酸(EGTA)等試劑。一般情況下��,提取液成分復(fù)雜��,需要對(duì)樣品中所含蛋白質(zhì)進(jìn)行富集和凈化�����。食品中蛋白質(zhì)富集凈化常用方法包括使用有機(jī)溶劑��、等電點(diǎn)沉淀蛋白和十二烷基硫酸鈉聚丙烯酰胺凝膠電泳(SDS-PAGE) 分離富集蛋白,根據(jù)檢測(cè)目的的不同選擇合適的方法��?����;瘖y品中的蛋白質(zhì)提取���、富集方法與食品基本一致���。需要注意的是��,食品中蛋白質(zhì)組成復(fù)雜���,高豐度蛋白質(zhì)或肽段可能會(huì)干擾低豐度蛋白質(zhì)或肽段的檢測(cè),需要有效的預(yù)分離程序降低樣品的復(fù)雜性�����。此外��,通過(guò)鳥(niǎo)槍法這種蛋白質(zhì)組學(xué)方法可將1 個(gè)蛋白質(zhì)分子轉(zhuǎn)化為多個(gè)肽段���,從而導(dǎo)致樣品復(fù)雜度增加[58]�����。在血液樣本中�����,利用高豐度蛋白質(zhì)去除試劑盒可以去除樣本中的高豐度干擾蛋白質(zhì)���,提高檢測(cè)痕量生物標(biāo)志物的靈敏度[59]��。目前�����,食品領(lǐng)域還未涉及高豐度蛋白質(zhì)去除試劑盒,主要利用凝膠電泳和液相色譜對(duì)蛋白質(zhì)或肽段進(jìn)行分離[60]���。同時(shí)�����,除提高提取效率外�����,還可從質(zhì)譜端入手�����,提高蛋白質(zhì)鑒定效率���,如所有理論碎片離子質(zhì)譜的順序窗口采集技術(shù)(SWATH-MS)使用相當(dāng)大的母離子分離窗口以系統(tǒng)且無(wú)偏的方式將給定樣品的所有離子化肽段落入指定質(zhì)量范圍內(nèi)��, 對(duì)所檢測(cè)的離子進(jìn)行全面的定性定量分析[61]��。
4.2 使用方法過(guò)程中的問(wèn)題與建議
近年來(lái)�����,隨著質(zhì)譜儀器性能的提升�����,質(zhì)譜法已被廣泛應(yīng)用于大分子蛋白質(zhì)和多肽的絕對(duì)定量研究領(lǐng)域��。其中�����,利用靶向蛋白質(zhì)組學(xué)技術(shù)��, 如多重反應(yīng)監(jiān)測(cè)(MRM)���、平行反應(yīng)監(jiān)測(cè)(PRM)技術(shù)結(jié)合標(biāo)準(zhǔn)品可以對(duì)目標(biāo)蛋白質(zhì)或者多肽進(jìn)行多重精確定量。利用基于質(zhì)譜的蛋白質(zhì)組學(xué)技術(shù)對(duì)蛋白質(zhì)含量進(jìn)行絕對(duì)定量分析時(shí)��,需要使用標(biāo)準(zhǔn)化參考樣品��,這是因?yàn)榇祟?lèi)研究間樣品比較在確定蛋白質(zhì)含量時(shí)所提供的信息只是相對(duì)的。目前��,蛋白質(zhì)組學(xué)技術(shù)絕對(duì)定量分析僅在有限數(shù)量的已知靶蛋白和肽上進(jìn)行�����,該技術(shù)主要受限于對(duì)合成和適當(dāng)表征的肽和蛋白質(zhì)標(biāo)準(zhǔn)品的需求��。一方面�����,對(duì)于加標(biāo)回收率的評(píng)價(jià)大多只涉及酶解前加入標(biāo)準(zhǔn)肽段���,回收率普遍較高,但只是評(píng)價(jià)了酶解過(guò)程的回收率�����,無(wú)法反映整個(gè)樣品前處理方法的真實(shí)回收率��,實(shí)際應(yīng)加入目標(biāo)蛋白來(lái)評(píng)定真實(shí)回收率�����。另一方面,肽段標(biāo)準(zhǔn)品定值方法多樣��,常用方法有質(zhì)量平衡法���、氨基酸分析法��、化學(xué)衍生化法和元素分析法等���,但這些方法普遍耗時(shí)長(zhǎng)、成本高��。有研究者開(kāi)發(fā)了基于定量標(biāo)簽特異性吸收紫外線(UV)波長(zhǎng)用于標(biāo)記肽的基于UV 的定量新方法��,該方法成本低���、準(zhǔn)確度高��,且能夠用于直接定量重組的UV 標(biāo)記肽���,直接控制和監(jiān)測(cè)與肽溶解度、沉淀以及吸附等有關(guān)的問(wèn)題[62]���。還有研究者使用高效液相色譜法(HPLC)檢測(cè)內(nèi)在酪氨酸熒光的替代方法��,表征了對(duì)應(yīng)合成的21殘基合成肽的熒光特性�����,并建立了一種涉及相對(duì)于非肽校準(zhǔn)物N-乙酰-L- 酪氨酸乙酯的定量方法[63]��。目前��,多肽標(biāo)準(zhǔn)物質(zhì)只有HPLC 級(jí)別��,純度僅大于95%��,不能準(zhǔn)確反映樣品中肽段含量���,建議相關(guān)部門(mén)盡快出品肽段標(biāo)準(zhǔn)物質(zhì)�����,或者由方法使用者自行標(biāo)定標(biāo)準(zhǔn)肽段�����。
參考文獻(xiàn)
[1] WALLS H, BAKER P, CHIRWA E, et al . Food security,food safety & healthy nutrition :Are they compatible?[J]. Glob Food Secur ,2019,21 :69-71.
[2] 全國(guó)人民代表大會(huì)常務(wù)委員會(huì). 中華人民共和國(guó)食品安全法(2021 年修正版)[EB/OL]. (2021-04-29).http://www.xxtq.gov.cn/xxtqsyj/zfxxgk/fdzdgknr/zcfg/zcfg_15120/t5092965.html.
[3] GAO Y, YE YW, XU JG, et al . Rapid and easy quantitative identification of Cronobacter spp. in infant formula milk powder by isothermal strand-exchange-amplification based molecular capturing lateral flow strip[J]. Food Contr ,2021,126(9761):108048.
[4] ZHANG H, WENG YX. Safety risks of plant fiber/plastic composites (PPCs) intended for food contact :A review of potential hazards and risk management measures[J]. Toxics ,2021,9(12):343.
[5] EFSA Panel on Biological Hazards (BIOHAZ Panel); KOUTSOUMANIS K, ALVAREZ-ORDÓÑEZ A, et al . The efficacy and safety of highpressure processing of food[J]. EFSA J ,2022,20(3):e07128.
[6] MOHAMMADI Z, JAFARI SM. Detection of food spoilage and adulteration by novel nanomaterial-based sensors[J]. Adv Colloid Interface Sci ,2020,286 :102297.
[7] 中國(guó)香料香精化妝品工業(yè)協(xié)會(huì). 化妝品行業(yè)“十四五”發(fā)展規(guī)劃[J]. 日用化學(xué)品科學(xué),2022,45(1):1-5,17.
[8] FIUME MM, BERGFELD WF, BELSITO DV, et al . Amended safety assessment of fatty acyl sarcosines and sarcosinate salts as used in cosmetics[J]. Int J Toxicol ,2021,40(2_suppl):117S-133S.
[9] 國(guó)家食品藥品監(jiān)督管理總局關(guān)于發(fā)布化妝品安全技術(shù)規(guī)范(2015 年版) 的公告[EB/OL]. (2015-12-23). https://www.nmpa.gov.cn/hzhp/hzhpfgwj/hzhpgzwj/20151223120001986.html.
[10] LEXMAULOVÁ H, GABROVSKÁ D, RYSOVÁ J, et al . ELISA kit for peanut protein determination :Collaborative study[J]. J AOAC Int ,2013,96(5):1041-1047.
[11] WINTERS AL, MINCHIN FR. Modification of the Lowry assay to measure proteins and phenols in covalently bound complexes[J]. Anal Biochem ,2005,346(1):43-48.
[12] HUESO D, FONTECHA J, GÓMEZ-CORTÉS P. Comparative study of the most commonly used methods for total protein determination in milk of different species and their ultrafiltration products[J]. Front Nutr ,2022,9 :925565.
[13] GU JC, XU CF, LI ML, et al . Species identification of silks from bombyx mori, eri silkworm and chestnut silkworm using Western blot and proteomics analyses[J]. Anal Sci ,2019,35(2):175-180.
[14] CHEN D, LI XY, ZHAO X, et al . Comparative proteomics of goat milk during heated processing[J]. Food Chem ,2019,275 :504-514.
[15] LIU GZ, DU YN, FU T, et al . Profiling protein interactions by purification with capillary monolithic affinity column in combination with label-free quantitative proteomics[J]. J Chromatogr A ,2022,1676 :463273.
[16] LI JP, ZHU HJ. Liquid chromatography-tandem mass spectrometry (LC-MS/MS)-based proteomics of drug-metabolizing enzymes and transporters[J]. Molecules ,2020,25(11):2718.
[17] NING T, SUN S, NIE J, et al . Simultaneous quantification of major capsid protein of human papillomavirus 16 and human papillomavirus 18 in multivalent human papillomavirus vaccines by liquid chromatography-tandem mass spectrometry[J]. J Chromatogr
A ,2020,1619 :460962.
[18] FIORINO GM, FRESCH M, BRÜMMER I, et al . Mass spectrometry-based untargeted proteomics for the assessment of food authenticity :The case of farmed versus wild-type salmon[J]. J AOAC Int ,2019,102(5):1339-1345.
[19] XIONG WL, PARKER CH, BOO CC, et al . Comparison of allergen quantification strategies for egg, milk, and peanut in food using targeted LC-MS/MS[J]. Anal Bioanal Chem ,2021,413(23):5755-5766.
[20] MULLER L, FORNECKER L, CIANFERANI S, et al . Tube-gel :A fast and effective sample preparation method for high-throughput quantitative proteomics[J]. Methods Mol Biol ,2019,1959 :123-127.
[21] GRIFFITHS RL, BERG JD. Automation of the whole-blood thiopurine S-methyltransferase (TPMT) phenotyping assay using the biomek NXP and biomek i5 liquid-handling workstations[J]. SLAS Technol ,2021,26(5):488-497.
[22] SHEN XJ, SUN LL. Systematic evaluation of immobilized trypsin-based fast protein digestion for deep and high-throughput bottomup proteomics[J]. Proteomics ,2018,18(9):e1700432.
[23] MESSNER CB,DEMICHEV V,WENDISCH D, et al . Ultra-high-throughput clinical proteomics reveals classifiers of COVID-19 infection[J].Cell Syst ,2020,11(1):11-24.e4.
[24] GALINDO-LUJÁN R, PONT L, MINIC Z, et al . Characterization and differentiation of quinoa seed proteomes by label-free mass spectrometry-based shotgun proteomics[J]. Food Chem ,2021,363 :130250.
[25] WANG JQ, XIAO J, LIU X, et al . Analysis of Tartary buckwheat (Fagopyrum tataricum) seed proteome using offline two-dimensional liquid chromatography and tandem mass spectrometry[J]. J Food Biochem ,2019,43(7):e12863.
[26] KOTECKA-MAJCHRZAK K, SUMARA A, FORNAL E, et al . Proteomic analysis of oilseed cake :A comparative study of species-specific proteins and peptides extracted from ten seed species[J]. J Sci Food Agric ,2021,101(1):297-306.
[27] WINDARSIH A, SURATNO, WARMIKO HD, et al . Untargeted metabolomics and proteomics approach using liquid chromatography-Orbitrap high resolution mass spectrometry to detect pork adulteration in Pangasius hypopthalmus meat[J]. Food Chem ,2022,386 :132856.
[28] HU LP, ZHANG HW, HU ZH, et al . Comparative proteomics analysis of three commercial tuna species through SWATH-MS based mass spectrometry and chemometrics[J]. Food Contr ,2022,141 :109162.
[29] PIOVESANA S, CAPRIOTTI AL, CARUSO G, et al . Labeling and label free shotgun proteomics approaches to characterize muscle tissue from farmed and wild gilthead sea bream (Sparus aurata)[J]. J Chromatogr A ,2016,1428 :193-201.
[30] ZHANG YD, MIN L, ZHANG S, et al . Proteomics analysis reveals altered nutrients in the whey proteins of dairy cow milk with different thermal treatments[J]. Molecules ,2021,26(15):4628.
[31] SUN XH, YU ZN, LIANG CZ, et al . Comparative analysis of changes in whey proteins of goat milk throughout the lactation cycle using quantitative proteomics[J]. J Dairy Sci ,2023,106(1):792-806.
[32] JI ZY, ZHANG JY, DENG CX, et al . Identification of mare milk adulteration with cow milk by liquid chromatography-high resolution mass spectrometry based on proteomics and metabolomics approaches[J]. Food Chem ,2023,405(Pt B):134901.
[33] LÓPEZ-PEDROUSO M, LORENZO JM, DI STASIO LD, et al . Quantitative proteomic analysis of beef tenderness of Piemontese young bulls by SWATH-MS[J]. Food Chem ,2021,356 :129711.
[34] BOUDON S, OUNAISSI D, VIALA D, et al . Label free shotgun proteomics for the identification of protein biomarkers for beef tenderness in muscle and plasma of heifers[J]. J Proteomics ,2020,217 :103685.
[35] ZHENG KZ, YIN YY, CAO Y, et al . Proteomic and parallel reaction monitoring approaches to evaluate biomarkers of mutton tenderness[J]. Food Chem ,2022,397 :133746.
[36] KANG WH, ZHANG JK, LI H, et al . Quantification of major allergens in peach based on shotgun proteomics using liquid chromatographytandem mass spectrometry[J]. LWT ,2022,160 :113234.
[37] TAO YX, YIN ST, FU LL, et al . Identification of allergens and allergen hydrolysates by proteomics and metabolomics :A comparative study of natural and enzymolytic bee pollen[J]. Food Res Int ,2022,158 :111572.
[38] STELLA R, SETTE G, MORESSA A, et al . LC-HRMS/MS for the simultaneous determination of four allergens in fish and swine food products[J]. Food Chem ,2020,331 :127276.
[39] GARCÍA-BÉJAR B, OWENS RA, BRIONES A, et al . Differential distribution and proteomic response of Saccharomyces cerevisiae and nonmodel yeast species to zinc[J]. Environ Microbiol ,2020,22(11):4633-4646.
[40] FU LL, WANG R, WANG YB, et al . Proteomic identification of responsive proteins of Vibrio parahaemolyticus under high hydrostatic pressure[J]. J Sci Food Agric ,2014,94(13):2630-2638.
[41] 烏日娜, 薛亞婷, 張平, 等. 豆醬微生物宏蛋白質(zhì)組提取及分析[J]. 食品科學(xué),2017,38(14):17-23.
[42] 國(guó)家藥監(jiān)局關(guān)于發(fā)布《已使用化妝品原料目錄(2021 年版)》的公告[EB/OL]. (2021-04-30). https://www.nmpa.gov.cn/xxgk/ggtg/hzhpggtg/jmhzhptg/20210430162707173.html.
[43] WANG T, YANG LT, WANG GF, et al . Biocompatibility, hemostatic properties, and wound healing evaluation of tilapia skin collagen sponges[J]. J Bioact Compatible Polym ,2021,36(1):44-58.
[44] JOHNSON W, BERGFELD WF, BELSITO DV, et al . Safety assessment of silk protein ingredients as used in cosmetics[J]. Int J Toxicol ,2020,39(3_suppl):127S-144S.
[45] LIN P, HUA N, HSU YC, et al . Oral collagen drink for antiaging :Antioxidation, facilitation of the increase of collagen synthesis, and improvement of protein folding and DNA repair in human skin fibroblasts[J]. Oxid Med Cell Longev ,2020,2020 :8031795.
[46] 司磊磊, 張燕, 侯虎, 等. 兩種魚(yú)皮膠原蛋白的比較及其降解物中多肽的識(shí)別[J]. 食品工業(yè)科技,2018,39(5):7-12.
[47] GUAN YX, HE JL, CHEN JD, et al . Valorization of fish processing by-products :Microstructural, rheological, functional, and properties of silver carp skin type Ⅰ collagen[J]. Foods ,2022,11(19):2985.
[48] RONG YH, ZHANG GN, WANG C, et al . Quantification of type Ⅰ and Ⅲ collagen content in normal human skin in different age groups[J]. Zhonghua Shao Shang Za Zhi ,2008,24(1):51-53.
[49] ZHANG Y, CHEN Y, ZHAO B, et al . Detection of Type Ⅰ and Ⅲ collagen in porcine acellular matrix using HPLC-MS[J]. Regen Biomater ,2020,7(6):577-582.
[50] KUMAZAWA Y, TAGA Y, IWAI K, et al . A rapid and simple LC-MS method using collagen marker peptides for identification of the animal source of leather[J]. J Agric Food Chem ,2016,64(30):6051-6057.
[51] 張雨彤, 魏夢(mèng)雅, 任倩倩, 等. 化妝品植物原料(Ⅵ):在抗衰老化妝品中的研究與開(kāi)發(fā)[J]. 日用化學(xué)工業(yè),2021,51(11):1052-1059.
[52] BURNETT CL, BOYER IJ, BERGFELD WF, et al . Safety assessment of plant-derived proteins and peptides as used in cosmetics[J]. Int J Toxicol , 2022, 41(Suppl.2):5S-20S.
[53] SGS 化妝品部. 歐盟通報(bào)化妝品修訂草案, 將限定花生蛋白等含量[J]. 日用化學(xué)品科學(xué),2017,40(6):14.
[54] SHARMA S, PRADHAN R, MANICKAVASAGAN A, et al . Corn distillers solubles by two-step proteolytic hydrolysis as a new source of plant-based protein hydrolysates with ACE and DPP4 inhibition activities[J]. Food Chem ,2023,401 :134120.
[55] COURTOIS J, BERTHOLET C, TOLLENAERE S, et al . Detection of wheat allergens using 2D Western blot and mass spectrometry[J]. J Pharm Biomed Anal ,2020,178 :112907.
[56] JIA HM, ZHOU TJ, ZHU H, et al . Quantification of Gly m 5.0101 in soybean and soy products by liquid chromatography-tandem mass spectrometry[J]. Molecules ,2018,24(1):68.
[57] ZHENG WM, YANG PY, SUN CY, et al . Comprehensive comparison of sample preparation workflows for proteomics[J]. Mol Omics ,2022,18(6):555-567.
[58] CHRIST D, WINTER G. Identification of protein domains by shotgun proteolysis[J]. J Mol Biol ,2006,358(2):364-371.
[59] LIU B, QIU FH, VOSS C, et al . Evaluation of three high abundance protein depletion kits for umbilical cord serum proteomics[J].Proteome Sci ,2011,9(1):24.
[60] LIU WJ, SONG QQ, CAO Y, et al . Advanced liquid chromatography-mass spectrometry enables merging widely targeted metabolomics and proteomics[J]. Anal Chim Acta ,2019,1069 :89-97.
[61] LUDWIG C, GILLET L, ROSENBERGER G, et al . Data-independent acquisition-based SWATH-MS for quantitative proteomics :A tutorial[J].Mol Syst Biol ,2018,14(8):e8126.
[62] SCHNATBAUM K, SOLIS-MEZARINO V, POKROVSKY D, et al . New approaches for absolute quantification of stable-isotope-labeled peptide standards for targeted proteomics based on a UV active tag[J]. Proteomics ,2020,20(10):e2000007.
[63] PRESTON GW, PHILLIPS DH. Quantification of a peptide standard using the intrinsic fluorescence of tyrosine[J]. Anal Bioanal Chem ,2016,408(9):2187-2193.






